蛋白质乙酰化: 真核细胞中的一种重要翻译后修饰
日期:2024-02-02 16:03:52
众所周知,翻译后修饰(PTM)是真核细胞实现蛋白质功能多样化和动态协调信号网络的重要机制。1964年,Vincent Allfrey及其同事首次发现了组蛋白上的赖氨酸乙酰化修饰。随后,以质谱仪为基础的蛋白质组学大大加速了内源性乙酰化蛋白质的发现和鉴定,同时也揭示了非组蛋白乙酰化的调控过程。乙酰化修饰是一种进化保守的PTM,存在于原核生物和真核生物中。下文将重点介绍蛋白质乙酰化的定义、功能、机制以及相关疾病。
1. 什么是蛋白质乙酰化?
蛋白质乙酰化是真核生物中主要的翻译后修饰(PTMs)之一,其中乙酰辅酶A(Ac-CoA)的乙酰基被引入多肽链上的特定位点 [1]。蛋白质被乙酰化的部位要么是各种氨基末端残基,要么是赖氨酸残基的ε-氨基。大多数真核生物蛋白质和调节肽在氨基末端残基上发生乙酰化,而赖氨酸乙酰化则发生在组蛋白和转录因子等不同蛋白质的不同位点上。
2. 蛋白质乙酰化的功能
人类的大多数蛋白质都会发生乙酰化。蛋白质的乙酰化可影响蛋白质的电荷、构象、稳定性、定位、合成以及与其他分子的相互作用。已发现许多乙酰化蛋白质参与了多种细胞过程,如翻译、转录、蛋白质折叠、细胞分裂、DNA损伤修复、信号转导和新陈代谢。组蛋白的乙酰化可降低其正电荷,削弱其与DNA结合的能力,并导致核小体解聚,从而使转录因子和RNA聚合酶顺利与DNA结合,激活基因的转录活性。研究发现,乙酰化可影响核酸酶的酶活性,从而调节底物RNA的水平 [7]。乙酰化还能调节100多种非组蛋白,包括转录因子、转录辅激活因子和核受体 [8]。非组蛋白乙酰化使细胞功能复杂化,而线粒体关键酶的乙酰化则调节生物能代谢。蛋白质乙酰化还与蛋白质降解有关。此外,蛋白质乙酰化还能调节多种信号通路并影响细胞周期。
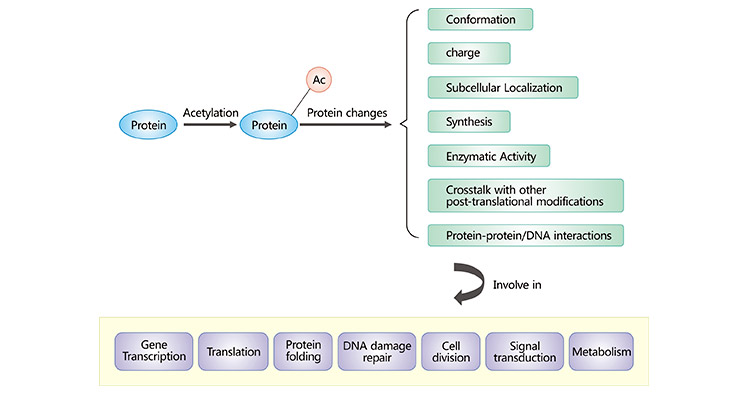
图1. 蛋白质乙酰化在真核细胞中的作用
3. 蛋白质乙酰化的机制
研究最多的蛋白质乙酰化发生在氨基上,但也检测到丝氨酸、苏氨酸和组氨酸残基上的乙酰化。蛋白质氨基的乙酰化有三种不同的机制:赖氨酸乙酰化(Nɛ-乙酰化)、蛋白质N端乙酰化(N α-乙酰化)和O-乙酰化。
3.1 Nɛ-赖氨酸乙酰化
赖氨酸乙酰化又称 Nɛ-赖氨酸乙酰化,是指将乙酰基从乙酰辅酶 A(乙酰辅酶 A)转移到蛋白质中赖氨酸侧链 ɛ 位的伯胺上。这一可逆过程中和了赖氨酸侧链ɛ位的正电荷。失去正电荷和增大的赖氨酸会破坏盐桥并引入立体结构,从而改变蛋白质-蛋白质/DNA 之间的相互作用、稳定性和酶活性 [2][3]。
有力的证据表明,蛋白质赖氨酸乙酰化可通过两种不同的机制发生:酶促乙酰化和非酶促乙酰化(化学乙酰化)[4]。两种不同的机制显示了对不同赖氨酸乙酰化位点的偏好,并揭示了这些赖氨酸位点相对乙酰化变化的不同动态。
酶促乙酰化依靠乙酰辅酶 A(Ac-CoA)乙酰转移酶催化乙酰基向赖氨酸残基的ɛ-氨基转移。非酶促乙酰化发生在乙酰供体和蛋白质之间。在真核细胞中,特别是在线粒体内,高能硫代酯类 AcCoA 可对蛋白质进行化学乙酰化 [16]。
3.2 Nα-乙酰化
Nα-乙酰化是指在N端氨基酸的 α-氨基上添加乙酰基。这是一个典型的不可逆过程,由 N-α-乙酰转移酶(NATs)介导。大约85%的人类蛋白质是通过 Nα-乙酰化修饰的 [5]。在真核生物中,Nα-乙酰化非常常见,而且是翻译过程中的共同作用,但在细菌中却很少见,而且是翻译后的作用 [17]。在真核细胞中,Nα-乙酰化要么发生在蛋氨酸的游离氨基上,要么发生在N端蛋氨酸裂解后暴露的氨基酸上。在细菌、线粒体和叶绿体中,Nα-乙酰化是翻译后进行的,因为蛋氨酸必须首先进行变形。
3.3 O-乙酰化
O-乙酰化是指在酪氨酸/丝氨酸/苏氨酸残基的羟基上添加乙酰基 [6]。它已被确定为蛋白质乙酰化的第三种类型。耶尔森氏菌外层蛋白J(YopJ)会对激活MAPK/ERK激酶和IκB激酶家族所需的丝氨酸和苏氨酸残基侧链进行乙酰化,从而阻断它们的磷酸化,抑制信号转导。蛋白质N-聚糖中的硅醛酸的O-乙酰化是一种重要的修饰,可以在 4-、7-、8-或9-位上以不同的组合出现。
4. 蛋白质乙酰化与疾病
功能蛋白的乙酰化和去乙酰化在胚胎发育、出生后成熟、心肌细胞分化、心脏重塑以及各种心血管疾病(包括肥胖、糖尿病、心脏代谢性疾病、缺血再灌注损伤以及心脏重塑、高血压和心律失常等)的发病过程中发挥着重要作用 [10] [11]。
欧阳洁等研究表明,肾小管上皮细胞线粒体中 SOD2 和 p53 蛋白乙酰化的升高是缺血再灌注(I/R)诱导急性肾损伤(AKI)发病机制中的一个重要信号事件 [9]。一些研究还表明,恢复 SIRT1/3 的活性可能是治疗 AKI 的新靶点。SIRT1 和 3 是依赖于烟酰胺腺嘌呤二核苷酸的蛋白去乙酰化酶,在抗氧化应激和抗细胞凋亡过程中发挥保护肾功能的作用。使用白藜芦醇可以有效恢复SIRT1/3的活性。研究表明,TDP-43的K136乙酰化会损害其RNA结合和剪接能力,促进病理磷酸化和泛素化TDP-43的不溶性聚集体的积累,这与肌萎缩性脊髓侧索硬化症(ALS)有关。
某些蛋白质的乙酰化与癌变有关。与相应的非肿瘤组织细胞相比,结肠癌相关转录因子1(CCAT1)在食管鳞状细胞癌(ESCC)细胞中的表达明显更高 [12]。众所周知,CCAT1的高表达可促进细胞增殖和侵袭,而下调 CCAT1 则可抑制这两个生物学过程 [13]。据报道,H3K27的乙酰化可部分上调CCAT1的表达,而CCAT1有可能诱发癌症 [14]。此外,糖酵解为癌细胞快速增殖提供了大量能量。磷酸甘油酸激酶1(PGK1)是糖酵解过程中的重要还原酶,如果发生乙酰化,可能会改变癌细胞的增殖。在肝癌细胞中,PGK1的活性会因乙酰化而增强,并进一步加速肿瘤细胞的增殖 [15]。
质谱技术与生物学的结合使大量乙酰化位点在所有蛋白质中的定位成为可能。越来越多的研究表明,乙酰化是自然界中最丰富的化学修饰之一,可能影响蛋白质的各种生理过程,甚至导致某些疾病。因此,蛋白质乙酰化也是近年来开发和设计治疗多种疾病的新药的一个很有前景的靶点。
5. 乙酰化相关产物
许多乙酰转移酶都参与了蛋白质的乙酰化过程。在此,CUSABIO为科研人员提供一些优质的乙酰转移酶,用于蛋白质乙酰化相关的研究。
| 乙酰化蛋白 | 乙酰转移酶 |
|---|---|
| Human proteins | NatA (catalytic subunit: Naa10, Naa15 ), NatB (Naa20, Naa25), NatC (Naa30, Naa35, Naa38), NatD (Naa40), NatE (Naa50), NatF (Naa60), NatH (Naa80) |
| Histones (H2A, H2B, H3, H4) | Gcn5, PCAF, Hat1, Elp3, Hpa2, Esa1, MOF, Sas2, Sas3, Tip60, MORF, TAFII250, TFIIIC, ACTR, and SRC1. |
| E. coli ribosomal proteins (S18, S5, and L12) | RimI, RimJ, RimL |
| Transcription factors (p53, E2F1-3, EKLF) | PCAF/Gcn5, p300/CBP, TAFII250 |
| Nuclear import factors (importin-α7 and Rch1) | p300/CBP |
参考文献:
[1] Adrian Drazica, Line M.Myklebust, et al. The world of protein acetylation [J]. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics Volume 1864, Issue 10, October 2016, Pages 1372-1401.
[2] David G. Christensen, Xueshu Xie, et al. Post-translational Protein Acetylation: An Elegant Mechanism for Bacteria to Dynamically Regulate Metabolic Functions [J]. Front Microbiol. 2019; 10: 1604.
[3] Ibraheem Ali, Ryan J. Conrad, et al. Lysine Acetylation Goes Global: From Epigenetics to Metabolism and Therapeutics [J]. Chem Rev. manuscript; available in PMC 2019 Jul 3.
[4] Miao-Miao Wang, Di You & Bang-Ce Ye. Site-specific and kinetic characterization of enzymatic and nonenzymatic protein acetylation in bacteria [J]. Sci Rep 7, 14790 (2017).
[5] Hollebeke J, Van Damme P and Gevaert K: N-terminal acetylation and other functions of Nalpha-acetyltransferases [J]. Biol Chem. 393:291–298. 2012.
[6] Yang XJ and Gregoire S. Metabolism, cytoskeleton and cellular signalling in the grip of protein Nepsilon- and O-acetylation [J]. EMBO Rep. 8:556–562. 2007.
[7] Song L, Wang G, et al. Reversible acetylation on Lys501 regulates the activity of RNase II [J]. Nucleic Acids Res. 2016;44:1979–1988.
[8] Narita T, Weinert BT, et al. Functions and mechanisms of non-histone protein acetylation [J]. Nat Rev Mol Cell Biol. 2019;20:156–174.
[9] Jie Ouyang, Zhenhua Zeng, et al. SIRT3 Inactivation Promotes Acute Kidney Injury Through Elevated Acetylation of SOD2 and p53 [J]. J Surg Res. 2019 Jan;233:221-230.
[10] Can Xia, Yu Tao, et al. Protein acetylation and deacetylation: An important regulatory modification in gene transcription (Review) [J]. Exp Ther Med. 2020 Oct; 20(4): 2923–2940.
[11] MingjieYang, YingmeiZhang, et al. Acetylation in cardiovascular diseases: Molecular mechanisms and clinical implications [J]. Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease Volume 1866, Issue 10, 1 October 2020, 165836.
[12] Hu M, Zhang Q, et al. lncRNA CCAT1 is a biomarker for the proliferation and drug resistance of esophageal cancer via the miR- 143/PLK1/BUBR1 axis [J]. Mol Carcinog. 2019;58:2207–2217.
[13] Li J and Qi Y. Ginsenoside Rg3 inhibits cell growth, migration and invasion in Caco-2 cells by downregulation of lncRNA CCAT1 [J]. Exp Mol Pathol. 2019;106:131–138.
[14] Zhang E, Han L, et al. H3K27 acetylation activated-long non-coding RNA CCAT1 affects cell proliferation and migration by regulating SPRY4 and HOXB13 expression in esophageal squamous cell carcinoma [J]. Nucleic Acids Res. 2017;45:3086–3101.
[15] Hu H, Zhu W, et al. Acetylation of PGK1 promotes liver cancer cell proliferation and tumorigenesis [J]. Hepatology. 2017;65:515–528.
[16] Hosp F, Lassowskat I, et al. Lysine acetylation in mitochondria: from inventory to function [J]. Mitochondrion 2017. 33:58–71.
[17] Soppa J. Protein acetylation in archaea, bacteria, and eukaryotes [J]. Archaea 2010:820681.
上一篇: 蛋白质磷酸化











