组蛋白SUMOylation
在2003年,Shiio和Eisenman首次在人类HEK293T和P493-6 B细胞中描述了组蛋白SUMOylation,并且显示其导致转录抑制 [1]。随后,组蛋白SUMOylation也在酵母 [2]、寄生原生动物 [3]和植物 [4]中得到报道。
1. 什么是组蛋白SUMOylation?
组蛋白SUMOylation是指小泛素样修饰剂(SUMO)与组蛋白蛋白质上的赖氨酸(K)残基共价结合的过程。
与组蛋白泛素化类似,所有核心组蛋白、连接组蛋白H1、组蛋白变体H2A.Z和H2A.X,以及着丝粒组蛋白变体Cse4在酵母中都可以发生SUMOylation [5-7]。在哺乳动物中,SUMOylation主要发生在H4上,H4的SUMOylation会引发转录抑制 [1,8]。由于缺乏像H3K9me和H3K27me这样的抑制性赖氨酸甲基化标记,酵母中SUMOylation目前是唯一已知的抑制性组蛋白标记。
2. 参与组蛋白SUMOylation的三个组成
组蛋白SUMOylation是一个可逆的过程,由几种酶和蛋白负责作为“写入者”、“读取者”和“擦除者”。
2.1 写入者
组蛋白SUMOylation写入者是负责将SUMO标记添加到组蛋白上的酶。
| SUMO 酶 | 物种 | 写入者 |
|---|---|---|
| E1 | 哺乳动物 | SAE1 (Aos1)/SAE2 (Uba2) |
| E2 | 酵母和高等真核生物 | Ubc9 |
| E3 | 酵母 | Siz1, Siz2 (Nfi1), and Mms21 |
| 高等真核生物 | PIAS family proteins: PIAS1, PIAS2, PIAS3, and PIAS4; RanBP2, Pc2/CBX4 |
2.2 读取者
组蛋白SUMOylation读取蛋白是能够识别和结合SUMO化的组蛋白,从而将信号转化为不同生物学效应的蛋白质。许多蛋白质具有特异的SUMO相互作用结构(SIMs),使它们能够结合到SUMO修饰的蛋白质,包括SUMO化的组蛋白。这些含有SIM的蛋白质可以包括转录因子、染色质重塑蛋白和其他调控蛋白。
| 读取者 | SUMOylated组蛋白 |
|---|---|
| HP1/CBX Proteins (HP1α/CBX5, HP1β/CBX1, and HP1γ/CBX3) | 结合SUMO化的组蛋白,特别是在异染色质区域;参与异染色质的维持 |
| TIF1 (TIF1α/TRIM24 and TIF1β/TRIM28) | 与SUMO修饰的组蛋白相互作用,发挥转录调控作用 |
| PML | 与SUMO修饰的组蛋白相互作用,有助于基因表达和基因组稳定性的调控 |
| SETDB1 | 在异染色质形成的背景下与SUMO化的组蛋白相互作用 |
| ATRX | 与SUMO修饰的组蛋白相互作用,并参与异染色质的调控 |
| BRCA1 | 在DNA损伤点识别SUMO化的组蛋白 |
| PCNA | 在DNA复制和修复过程中与SUMO修饰的组蛋白相互作用 |
| BRD4 | 在转录调控的背景下与SUMO修饰的组蛋白相互作用 |
2.3 擦除者
组蛋白SUMOylation可以通过去SUMO化酶进行逆转,也称为擦除者,其中包括特异性蛋白酶——泛素样蛋白酶(SENP),它能够去除组蛋白蛋白质上的SUMO化标记。
3. 组蛋白SUMOylation机制
组蛋白SUMOylation通过多步骤的酶依赖性反应协同进行,涉及SUMO E1激活酶、E2连接酶和E3连接酶。所有SUMO蛋白,包括SUMO1-4,首先被催化成成熟形式,其中C-末端的二甘氨酸对于E1介导的腺苷酸化是必不可少的。E1酶,由SAE1和SAE2亚基组成的异源二聚体,以ATP依赖的方式触发SUMO蛋白的激活,形成E1-SUMO中间体。一旦被激活,SUMO随后通过酯交换反应从E1-SUMO中间体转移到E2连接酶UBC9上。最后,E3连接酶PIAS(活化STAT的蛋白抑制剂)和RanBP2(Ran结合蛋白2)促使SUMO从UBC9转移到目标组蛋白上的特定赖氨酸残基。
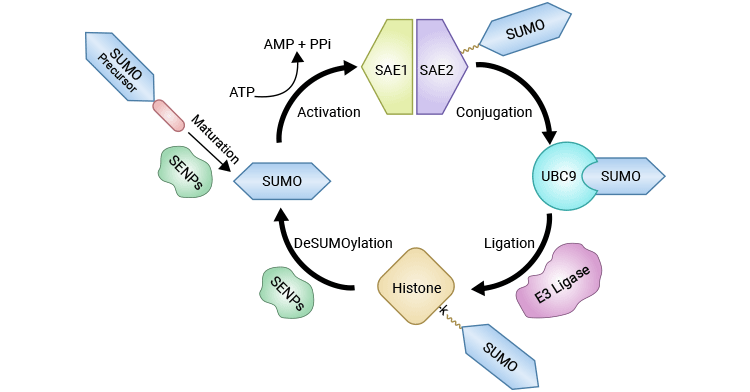
图 1. 组蛋白SUMOylation和deSUMOylation的催化循环
该图片引自:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9654019/
4. 组蛋白SUMOylation的功能
组蛋白SUMOylation在共转录过程中具有多种功能,包括转录调控、染色质重塑、转录延伸和阻止隐性起始。
组蛋白SUMOylation可以导致核小体结构的改变,从而进行染色质重塑,并影响各种转录因子与DNA的结合,从而抑制某些基因的转录。
多项研究表明,组蛋白SUMOylation导致转录活性的降低。例如,组蛋白4的SUMOylation通过招募HDACs和异染色质蛋白1(HP1)介导转录抑制。有趣的是,最近的研究表明,SUMOylation调节了组蛋白修饰酶的活性,例如HDAC1、HDAC2、HDAC4、SIRT1、EZH2和KDM5B,从而间接调节染色质状态。
此外,在胚胎干细胞中,链霉亲和素2/3(SUMO2/3)修饰连接器组蛋白H1促使其结合到高度致密的异染色质中。相反,缺乏SUMOylation会导致染色质解紧缩和全能性的恢复 [9]。
表 1. 组蛋白SUMOylation位点和功能
| 物种 | 组蛋白 | SUMOylated位点 | 功能 |
|---|---|---|---|
| Human | H2A | - | 转录抑制或染色质压缩 |
| H3 | K18 | - | |
| H4 | K12 | - | |
| H2AX | K5, K9, K13, K15, K118, K119, K127, K133, K134 | - | |
| S. cerevisiae | H2A | K126 | 转录抑制/激活,抑制潜在启动 |
| H2B | K6, K7, K16, K17 | - | |
| H3 | - | ||
| H4 | K5, K8, K12, K16, K20 | - | |
| H2A.Z | K126, K133 | DNA双链断裂修复 | |
| Cse4 | K65, K215, K216 | Cse4的整合或蛋白质降解 |
5. 组蛋白SUMOylation与其他组蛋白修饰的相互作用
H4的SUMOylation增加了其与组蛋白去乙酰化酶HDAC1和异染色质蛋白1 HP1γ的相互作用,与SUMO在转录抑制中的作用一致 [1]。已经证明SUMO对于沉积在与基因沉默相关的异染色质的H3K9me3分子标志是必需的 [10,11]。
Nathan及其团队巧妙地运用了化学、生化和遗传方法,提供了令人信服的证据,表明组蛋白SUMOylation对它们的乙酰化起对抗作用,确立了它作为一种在进化上保守的抑制性标记 [4]。在酿酒酵母中,已经证明组蛋白SUMOylation对抗某些激活修饰,如乙酰化和H2B泛素化(可能在相同的赖氨酸残基上)[4]。Chatterjee实验室发现H4K12su能够刺激转录抑制性的LSD1-CoREST1-HDAC1复合物中的去甲基化酶和去乙酰化酶活性。
许多被SUMO化的赖氨酸可以被甲基化、乙酰化或泛素化。在细胞周期中,SUMOylation与磷酸化之间发生相互作用,以实现细胞周期的调节。
6. 组蛋白Sumoylation与疾病
越来越多的证据表明,组蛋白SUMOylation在胚胎发育、细胞分化和疾病状态中发挥着关键作用。SUMOylation过程的失调与各种癌症、神经退行性疾病和免疫性疾病有关。组蛋白SUMOylation对于维持组蛋白功能和DNA转录非常重要,其失衡将影响细胞周期、分化和凋亡,并可能导致肿瘤的发展。
参考文献:
[1] Shiio Y, Eisenman RN. 2003 Histone sumoylation is associated with transcriptional repression [J]. Proc. Natl Acad. Sci. USA 100, 13 225-13 230.
[2] Ryu, H. Y., Su, D., et al. (2019) The Ulp2 SUMO protease promotes transcription elongation through regulation of histone sumoylation [J]. EMBO J. 38, e102003.
[3] Issar, N., Roux, E., Mattei, D., and Scherf, A. (2008) Identification of a novel post-translational modification in Plasmodium falciparum: protein sumoylation in different cellular compartments [J]. Cell. Microbiol. 10, 1999–2011.
[4] Miller, M. J., Barrett-Wilt, et a. (2010) Proteomic analyses identify a diverse array of nuclear processes affected by small ubiquitin-like modifier conjugation in Arabidopsis [J]. Proc. Natl. Acad. Sci. U.S.A. 107, 16512–16517.
[5] Nathan, D., Ingvarsdottir, K., et a. (2006) Histone sumoylation is a negative regulator in Saccharomyces cerevisiae and shows dynamic interplay with positive-acting histone modifications [J]. Genes Dev. 20, 966– 976.
[6] Kalocsay M, Hiller NJ, Jentsch S (2009) Chromosome-wide Rad51 spreading and SUMO-H2A.Z-dependent chromosome fixation in response to a persistent DNA double-strand break [J]. Mol Cell 33: 335–343.
[7] Ohkuni K., Takahashi Y., F, et a. SUMO-targeted ubiquitin ligase (STUbL) Slx5 regulates proteolysis of centromeric histone H3 variant Cse4 and prevents its mislocalization to euchromatin [J]. Mol. Biol. Cell. 2016; 27:1500–1510.
[8] Shiio, Y., and Eisenman, R.N. (2003). Histone sumoylation is associated with transcriptional repression [J]. Proc Natl Acad Sci U S A 100, 13225–30.
[9] Sheban, D., Shani, T., et a. (2022). SUMOylation of linker histone H1 drives chromatin condensation and restriction of embryonic cell fate identity [J]. Mol. Cell. 82, 106–122.e9.
[10] Ninova M, Godneeva B, et al. 2020 The SUMO Ligase Su(var)2–10 controls hetero- and euchromatic gene expression via establishing H3K9 trimethylation and negative feedback regulation [J]. Mol. Cell 77, 571-585.e4.
[11] Ninova M, Fejes Toth K, Aravin AA. 2019 The control of gene expression and cell identity by H3K9 trimethylation [J]. Development 146, dev181180.






