组蛋白泛素化
1. 什么是组蛋白泛素化?
将一个由76个氨基酸组成的泛素(Ub)部分连接到组蛋白的赖氨酸残基上的过程被称为组蛋白泛素化。组蛋白泛素化包括组蛋白单泛素化和组蛋白多泛素化,具体取决于连接到组蛋白上的泛素(Ub)部分的数量。组蛋白,特别是H2A和H2B,主要发生单泛素化,这是一个可逆的过程,改变了组蛋白的质量并影响了核小体的动态。然而,组蛋白多泛素化生成了一个不可逆的信号,用于蛋白酶体介导的降解。
在大多数较高等的真核生物中,约5%-15%的总H2A蛋白被泛素化,而H2B泛素化水平占1%-1.5% [1,2]。在酵母菌Saccharomyces cerevisiae中,约有10%的H2B被泛素化,而H2A的泛素化水平无法检测到 [2,3]。
组蛋白H2A主要在赖氨酸119(H2AK119ub1)处被泛素化,而H2B的主要泛素化位点是在酵母菌中的赖氨酸123(H2BK123ub1),在哺乳动物中的是赖氨酸120(H2BK120ub1)[1,2]。染色质免疫沉淀(ChIP)实验显示,单泛素化的H2A主要集中在基因组的卫星区域,而H2Bub则定位在活跃基因的体部。
除了H2A和H2B,泛素修饰还已经在核心组蛋白H3、H4和连接组蛋白H1上被报道。例如,在紫外线照射后,已经发现CUL4-DDB-RBX1泛素连接酶复合物在体内对H3和H4进行多泛素化 [4]。
2. 组蛋白泛素化的机制
泛素首先与E1酶结合,并通过在其C端甘氨酸和E1的活性半胱氨酸残基之间形成硫酯键的方式以ATP依赖的方式被激活。接下来,激活的泛素通过硫酯键与泛素结合的方式转移到E2酶。最后,E2与一个E3泛素连接酶相互作用,该酶将泛素共价连接到组蛋白的目标赖氨酸残基上。E3泛素连接酶的功能是将E2-泛素复合物定位到目标赖氨酸附近。
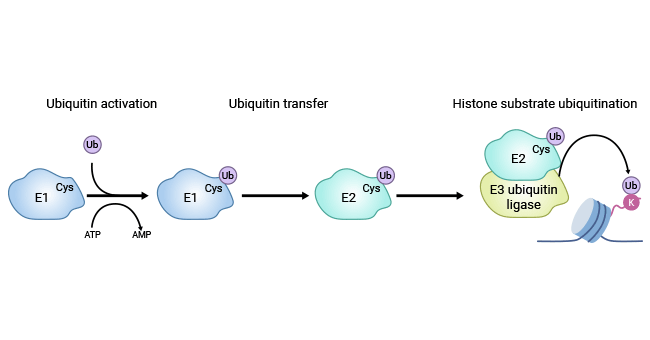
图1. 组蛋白单泛素化的过程
3. 组蛋白泛素化酶
组蛋白泛素化是一个经过精密调控的过程,涉及E1激活酶、E2结合酶和E3连接酶。这些步骤最终导致泛素(Ub)与组蛋白蛋白上的一个赖氨酸(Lys)残基形成共价键。
组蛋白泛素化是一个可逆的过程。去泛素化酶(DUBs)从泛素化的组蛋白上的赖氨酸残基上去除泛素。
| 酶 | 物种 | 组蛋白特异性 | 酶活 | 在转录中的作用 |
|---|---|---|---|---|
| RING1A/RING1B/BMI1 | Human | H2A | E3 | 抑制 |
| 2A-HUB | Human | H2A | E3 | 抑制 |
| BRCA1/BARD1 | Human | H2A | E3 | 抑制 |
| UbcH5c | Human | H2A | E2 | N/A |
| Bre1 | Yeast | H2B | E3 | 激活 |
| Rad6 | Yeast | H2B | E2 | 激活 |
| RNF20/RNF40 | Human | H2B | E3 | 激活 |
| RAD6A/RAD6B | Human | H2B | E2 | 激活 |
| UbcH6 | Human | H2B | E2 | 激活 |
| USP16 | Human | H2A | DUB | 激活 |
| USP21 | Human | H2A | DUB | 激活 |
| 2A-DUB | Human | H2A | DUB | 激活 |
| BAP1 | Human/Drosophila | H2A | DUB | 激活 |
| Ubp8 | Yeast | H2B | DUB | 激活 |
| Ubp10 | Yeast | H2B | DUB | 抑制 |
| Ubp7 | Drosophila | H2B | DUB | 抑制 |
| SCNY | Drosophila | H2B | DUB | 抑制 |
| UBP12/UBP46 | Xenopus | H2A H2B | DUB | 激活 |
| USP3 | Human | H2A H2B | DUB | N/A |
| USP22 | Human | H2A H2B | DUB | 激活 |
| BAP1 | Human/Drosophila | H2A | DUB | 激活 |
| Ubp8 | Yeast | H2B | DUB | 激活 |
表1: 单泛素化组蛋白修饰酶的功能。
该信息引用自:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3355875/
与其他类型的组蛋白修饰读取因子不同,泛素化组蛋白读取因子之间没有共享的结构域或结构模体。
| 修饰 | 读取蛋白 |
|---|---|
| H2AK119ub1 | PRC1, PRC2, RSF1, DNMT3A, ZRF1 |
| H2AK13/15ub1 | 53BP1, RNF169, RAD18, BARD1 |
| H2AK127/129ub1 | SMARCAD1, USP48 |
| H2BK120ub1 | Dot1L, COMPASS, MLL complexes, FACT, SWI/SNF, Chd1 |
| H2BK18/23ub1 | DNMT1 |
| H3K14ub1 | Clr4/SUV39H1 |
| H3K23/36/37ub1 | Gcn5 |
表2:组蛋白泛素化位点及相应的读取蛋白。
该表格信息来源于:https://www.frontiersin.org/articles/10.3389/fcell.2022.968398/full
4. 组蛋白泛素化的功能
由于组蛋白是最常见的泛素化蛋白,它们的泛素化在几乎所有DNA相关过程中扮演着关键角色,例如转录的启动和延伸、维持染色质结构、DNA复制和DNA修复。这些多样的影响主要取决于与染色质相关的组蛋白泛素化的位点特异性识别因子。
单泛素化组蛋白可以引发基因的激活和抑制,而多泛素化则为它们标记了蛋白酶体介导的降解。
H2A119ub1 调节组织特异性位点、非活性女性X染色体上的基因抑制,或者在DNA损伤区域。H2AK119泛素化还参与了连接组蛋白H1结合到核小体上 [5],Polycomb沉默 [6] 和雌性哺乳动物X染色体失活 [7]。
H2Bub1 参与转录激活和延伸、DNA损伤应答(DDR)和干细胞可塑性。在酿酒酵母和人类细胞中,H2Bub1 是H3K4和H3K79甲基化的前提条件 [8,9]。研究表明,H2Bub1 直接增强了DOT1L甲基转移酶的活性,从而促使了酵母和人类模型中H3K79甲基化的促进。
组蛋白泛素化还参与了精子形成,并在精子发生过程中起到了去除核小体的重要作用。
5. 组蛋白泛素化与其他组蛋白修饰的相互作用
组蛋白泛素化与其他组蛋白修饰之间的相互作用构成了一个复杂的调节网络,统治着染色质动态和基因表达。
已经证明组蛋白赖氨酸甲基化可以被组蛋白泛素化或组蛋白泛素化修饰酶交叉调控。单泛素化的H2A被PRC2识别,引发H3K27甲基化 [13]。这会招募SETDB1进行H3K9三甲基化,锚定染色质紧缩蛋白,如HP1 [14,15]。三甲基化的H3K27会吸引更多的PRC1,通过H2A修饰开始稳定的远程信号传递 [16]。H2B的单泛素化触发DOT1L对组蛋白H3的K79位点进行甲基化,随后通过MLL增强H3K4甲基化,促使转录激活因子的招募 [17]。
组蛋白泛素化还与DNA损伤应答中的赖氨酸乙酰化相互作用。敲除赖氨酸乙酰转移酶KAT5(TIP60)或PCAF导致H2B的赖氨酸120位点(H2BK120ac)的乙酰化减少,伴随着H2BK120的泛素化增加,以应答DNA损伤 [10]。EP300对H3K9me2脱甲基化酶JMJD1A进行乙酰化,阻止了STUB1介导的多泛素化,从而保持了JMJD1A水平 [11]。H2B的单泛素化还与H3甲基化相互作用,促进基因转录和端粒沉默。
在染色质控制中,泛素化与磷酸化之间存在相互关系。EGFR通过RNF8-UBE2L6使赖氨酸48-链接的多泛素化链附着到H3K4,导致H3的降解 [12]。当H3.3的苏氨酸11(H3.3T11ph)突变为丙氨酸时,磷酸化和泛素化均被阻断。在由电离辐射(IR)引发的DNA损伤信号传导过程中,组蛋白磷酸化和泛素化相互协同作用。
6. 组蛋白泛素化与疾病
组蛋白泛素化或去泛素化的失调被视为各种人类疾病的致病因素,包括与大脑有关的疾病和癌症相关的疾病。
影响调节H2Aub水平的各个组分的突变与多种综合征和大脑相关的障碍相关。例如,AUTS2基因内的突变与自闭症和智力障碍相关。PHC1的缺陷与大脑发育障碍相关,例如常染色体隐性原发性小头症。与H2A泛素化水平受损相关的PHC1突变与细胞周期进展受损和DNA修复机制受损相关。
PRC1的丧失与生命不相容。干扰PRC1活性的突变导致小头症或学习障碍。PRC1的异常调节还导致多个器官(包括大脑、肝脏、结肠、乳腺、肺、前列腺或淋巴系统)的肿瘤。
H2Bub1的异常单泛素化通常与肿瘤发生相关。RNF20/40在原发性肿瘤(如结肠、乳腺、卵巢、前列腺和肺癌)中常常受到干扰。H2Bub1的缺乏通常与癌症患者的不良预后相关 [18]。
环指蛋白和BMI1的扩增或过度表达导致H2Aub水平升高和肿瘤进展。位于BAP1的UCH、CTD和NLS功能结构域内的癌症突变导致H2Aub的去泛素化丧失,促使癌症的发展。
参考文献:
[1] Goldknopf I. L., Taylor C. W., et al. (1975). Isolation and characterization of protein A24, a “histone-like” non-histone chromosomal protein [J]. J. Biol. Chem. 250, 7182–7187.
[2] West M. H., Bonner W. M. (1980). Histone 2B can be modified by the attachment of ubiquitin [J]. Nucleic Acids Res. 8, 4671–4680.
[3] Robzyk K., Recht J., Osley M. A. (2000). Rad6-dependent ubiquitylation of histone H2B in yeast [J]. Science 287, 501–504.
[4] Wang H., Zhai L., et al. (2006). Histone H3 and H4 ubiquitylation by the CUL4-DDB-ROC1 ubiquitin ligase facilitates cellular response to DNA damage [J]. Mol. Cell 22, 383–394.
[5] Jason LJ, Finn RM, et al. Histone H2A ubiquitylation does not preclude histone H1 binding, but it facilitates its association with the nucleosome [J]. J. Biol. Chem. 2005;280:4975–4982.
[6] Wang H, Wang L, et al. Role of histone H2A ubiquitylation in Polycomb silencing [J]. Nature. 2004;431:873–878.
[7] Fang J, Chen T, et al. Ring1b-mediated H2A ubiquitylation associates with inactive X chromosomes and is involved in initiation of X inactivation [J]. J. Biol. Chem. 2004;279:52812–52815.
[8] Kim J, Guermah M, et al. RAD6-mediated transcription-coupled H2B ubiquitylation directly stimulates H3K4 methylation in human cells [J]. Cell. 2009;137:459–471.
[9] Nakanishi S, Lee JS, et al. Histone H2BK123 monoubiquitylation is the critical determinant for H3K4 and H3K79 trimethylation by COMPASS and Dot1 [J]. J. Cell Biol. 2009;186:371–377.
[10] Kim J.J. et al. Systematic bromodomain protein screens identify homologous recombination and R-loop suppression pathways involved in genome integrity [J]. Genes Dev. 2019; 33: 1751-1774.
[11] Xu S. et al. p300-mediated acetylation of histone demethylase JMJD1A prevents its degradation by ubiquitin ligase STUB1 and enhances its activity in prostate cancer [J]. Cancer Res. 2020; 80: 3074-3087.
[12] Xia Y. et al. RNF8 mediates histone H3 ubiquitylation and promotes glycolysis and tumorigenesis [J]. J. Exp. Med. 2017; 214: 1843-1855.
[13] Kalb R, Latwiel S, et al. (2014) Histone H2A monoubiquitylation promotes histone H3 methylation in Polycomb repression [J]. Nat Struct Mol Biol 21: 569–571.
[14] Zhao D, Zhang X, et al. (2016) The BAH domain of BAHD1 is a histone H3K27me3 reader [J]. Protein Cell 7: 222–226.
[15] Machida S, Takizawa Y, et al. (2018) Structural basis of heterochromatin formation by human HP1 [J]. Mol Cell 69: 385–397.e8.
[16] Tamburri S, Lavarone E, et al. (2020) Histone H2AK119 mono-ubiquitylation is essential for polycomb-mediated transcriptional repression [J]. Mol Cell 77: 840–856.e5.
[17] Wu L, Zee BM, et al. (2011) The RING finger protein MSL2 in the MOF complex is an E3 ubiquitin ligase for H2B K34 and is involved in crosstalk with H3 K4 and K79 methylation [J]. Mol Cell 43: 132–144.
[18] Sa Zhou, Yuqiao Cai, et al. Role of H2B mono-ubiquitylation in the initiation and progression of cancer [J]. Bulletin du Cancer, Volume 108, Issue 4, April 2021, Pages 385-398.






